Tabela de conteúdos
Biologia Molecular
Resumo geral
A biologia molecular é tratada como ferramenta do profissional envolvido, afim de esclarecer, até um determinado limite, suas perguntas. Além disso, a biologia molecular se apropriou de várias outras disciplinas e técnicas para a formação de seu escopo, de forma que todo esse corpo de conhecimento é praticamente imensurável e, dessa forma, é difícil de ser definido. Para uma busca mais atual desta disciplina é necessário que desfrutemos de conceitos como genoma e genética, que precisam ser delineados.
Genoma pode ser definido como a sequência completa de DNA de um organismo, logo, o conjunto de todos os genes. Estes, por sua vez, são definidos como trechos do DNA, ou seja, uma sequência de bases nitrogenadas (nucleotídeos) que carregam as informações necessárias para produção de proteínas [1]. A identificação genômica possibilita detectar diferenças genéticas entre as espécies ou entre indivíduos da mesma espécie. Desta forma, a análise do DNA para a identificação dos indivíduos baseia-se no fato de que cada ser humano tem sua composição genética única.
A compreensão de conceitos como replicação do DNA, transcrição e transcrição reversa e síntese proteica também se fazem necessários para o entendimento geral da biologia molecular e, precisam ser definidos. Replicação do DNA é um processo de duplicação das informações genéticas que estão presentes na molécula de DNA parental que é dividida em duas hélices que servem como molde para a formação de dois novos pares de moléculas de DNA[2] que são, por sua vez, cópias quase idênticas às parentais. Tais diferenças são geradas por possíveis mutações que ocorram no processo.
A replicação é considerada um processo semi-conservativo pois, cada nova dupla hélice mantém a metade originária da molécula parental. O processo de transcrição é a síntese de uma fita de RNA a partir de uma molécula de DNA como molde, assim, o RNA é a cópia de uma das duas fitas do DNA. Sendo este processo de extrema importância, já que é através dele que as informações genéticas são perpetuadas. Porém, oque ocorre na transcrição reversa é exatamente o contrário, onde, a síntese de moléculas de DNA acontecem a partir do RNA, logo, a informação genética também será perpetuada por meio deste processo. No entanto, a transcrição reversa ocorre somente nos chamados retrovírus, enquanto a transcrição aparece nos procariontes e eucariontes. [3].
A síntese proteica, também conhecida como tradução, é a produção de proteínas que promovem a manutenção e o crescimento da célula. Esse processo é dado em três etapas: iniciação da tradução, alongamento da cadeia polipeptídica e término da tradução; e acontece tanto em células procariontes quanto eucariontes[4].
Genoma e Genética
Definição de Genoma
O genoma é o conjunto de genes responsáveis por controlar os processos vitais das células, ou seja, nesse conjunto contém toda a informação que será passada, de forma hereditária, para os seus descendentes [5].
De uma forma mais geral, “os genes são unidades biológicas que determinam as características de um organismo” [6]. A quantidade de genes no genoma varia de indivíduo para indivíduo, por exemplo, uma bactéria simples possui apenas 500 genes, já os humanos possuem cerca de 30 mil [7]
Existem dois tipos de ácidos nucléicos capazes de armazenar os genes: o ácido desoxirribonucleico (DNA) e o ácido ribonucleico (RNA) [4].Esses ácidos são constituídos por nucleotídeos, esses nucleotídeos, por sua vez, são formados por um ácido fosfórico, uma pentose e uma base nitrogenada. Além disso, nas células eucariontes, eles se encontram no núcleo, tanto o DNA como o RNA, no entanto, uma das diferenças entre esses dois ácidos é que no caso do RNA e também pode estar presente no citoplasma [8].
Projeto Genoma Humano
A necessidade de se acelerar a compreensão dos mecanismos responsáveis pelo câncer e também pelo fato de querer estudar as alterações no DNA dos sobreviventes da bomba atômica fez com que, formalmente, em 1990, nos Estados Unidos, surgisse a ideia de se mapear os genes e sequenciar o genoma, essa tarefa foi denominada como Projeto Genoma Humano (PGH). [9]
Após sete anos, os resultados finais foram publicados, em abril de 2003, com 99% do genoma humano sequenciados e 99,99% de precisão. [10]
A ideia surgiu nos Estados Unidos, de forma bastante sistemática e a princípio foi sustentada basicamente pelo National Institutes of Health (NHI) e pelo Departamento de Energia (DOE), no entanto, esse projeto logo se tornou internacional, com o apoio de diversos países, incluído o Brasil.[9]
O geneticista norte-americano James Watson , ficou responsável pela coordenação inicial, contando com a participação de 5 mil cientistas de 18 países em 250 laboratórios. [11].
As principais agências que colaboraram, do Brasil, foram: a Fundação de Amparo à Pesquisa do Estado de São Paulo (FAPESP) , o Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq) e também o Programa de Apoio ao Desenvolvimento Científico e Tecnológico (PADCT) [9].
As principais metas do Projeto Genoma Humano foram:[10]
• Identificar todos os genes humanos;
• Determinar a sequência dos cerca de 3,2 bilhões de pares de bases que compõem o genoma do Homo sapiens;
• Armazenar a informação em bancos de dados;
• Desenvolver ferramentas de análise dos dados;
• Transferir a tecnologia relacionada ao Projeto para o setor privado;
• Colocar em discussão os problemas éticos, legais e sociais que pudessem surgir com o Projeto.
A seguir, alguns resultados obtidos na primeira publicação da sequência: [10]
• O genoma humano contem 3,2 bilhões de nucleotídeos;
• O tamanho médio dos genes é de 3.000 bases, mas varia muito, sendo o maior deles o gene da
distrofina com 2,4 milhões de pares de bases;
• A função de cerca de 50% dos genes descobertos é desconhecida;
• A sequência do genoma humano é 99,9% exatamente a mesma em todas as pessoas;
• Cerca de 2% do genoma codifica instruções para a síntese de proteínas;
• Sequências repetidas que não codificam proteínas constituem mais do que 50% do genoma humano;
• Não são conhecidas as funções para as sequências repetidas, mas elas ajudam a entender a
estrutura e a dinâmica dos cromossomos. Através dos anos essas repetições reformulam o genoma
rearranjando-o, criando desse modo genes inteiramente novos ou modificando genes já existentes;
• O genoma humano possui mais sequências repetidas (50%) do que Arabdopsis thaliana (11%), o
verme C. elegans (7%), e Drosophila (3%);
• Mais do que 40% das proteínas humanas preditas compartilham similaridade com as proteínas de
moscas e de vermes;
• Genes estão concentrados em áreas, ao acaso, ao longo do genoma, com vastas sequências sem
código para proteínas entre eles;
• O cromossomo 1 (o maior do genoma humano) tem o maior número de genes - 3.168 – e o
cromossomo Y, o menor -344;
• Algumas sequências gênicas específicas foram associadas com numerosas doenças e disfunções,
incluindo câncer de mama, doenças musculares, surdez e cegueira;
• Os cientistas localizaram, no genoma humano, milhares de locais nos quais há diferença de apenas
uma base. Essa informação promete revolucionar o processo de encontrar sequências de DNA
associadas com doenças muito comuns tais como disfunções cardiovasculares, diabetes, artrite e
câncer.
Definição de Genética
Genética é a área da biologia que se estudam os genes, analisa a forma como a hereditariedade biológica é transmitida pelas gerações e como é efetuado o desenvolvimento das características que controlam os respectivos processos.[12]
O termo Genética vem do grego genno, que significa “fazer nascer”, o conceito também faz referência àquilo que pertence ou que é relativo à génese ou origem das coisas[13].
A genética subdivide-se em vários ramos:
- Genética Clássica ou mendeliana;
- Genética Quantitativa;
- Genética Molecular;
- Genética de populações e evolutiva;
- Genética do desenvolvimento.
Genética Clássica
A história da genética, teve inicio em 1866, à partir da obra do frade agostiniano Gregor Johann Mendel. . Hoje, é considerado pai da genética clássica, com seu famoso experimento com as ervilhas, que veio a ser conhecido como Herança Mendeliana.
Mendel estabeleceu os padrões de hereditariedade de algumas características existentes em ervilhas, mostrando que obedeciam a regras estatísticas simples. Seu trabalho teve reconhecimento, apenas após sua morte, por cientistas que estavam trabalhando em projetos similares.
O ano de 1900 marcou a “Redescoberta de Mendel” por Hugo de Vries, Carl Correns. e Erich von Tschermak, e em 1915 os princípios básicos da genética mendeliana foram aplicados a uma grande variedade de organismos, mais notavelmente a mosca Drosophila melanogaster.[14]
Liderado por Thomas Hunt Morgan e seus colegas, os geneticistas desenvolveram o modelo mendeliano, que foi amplamente aceito. Juntamente com o trabalho experimental, matemáticos desenvolveram o quadro estatístico da genética de populações, trazendo explicações genéticas para o estudo da evolução.[14]
Após seus experimentos Mendel escreveu as 3 leis da hereditariedade, sendo elas os principais pilares da genética clássica. Nos dias atuais, algumas ideias da genética clássica foram abandonadas ou reformuladas de forma que façam sentido com a genética molecular/moderna. [15]
Experimento de Mendel
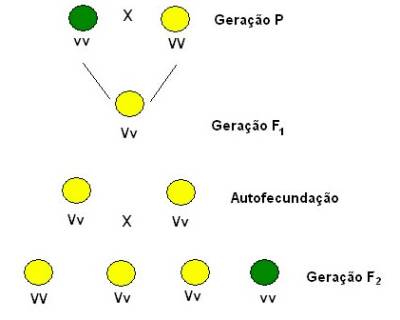
Mendel escolheu ervilhas-de-cheiro Pisum sativum, por ser de fácil cultivo, realizar autofecundação, possuir um ciclo reprodutivo curto e alta produtividade.
Utilizou como metodologia, o cruzamento entre linhagens de ervilhas “puras”, sendo consideradas puras, as ervilhas, que após seis gerações apresentavam as mesmas características. Realizou o cruzamento de polinização cruzada, que consistia na retirada do pólen de uma planta com semente amarela e depositá-lo sob o estigma de uma planta com sementes verdes.[16].
As características observadas por Mendel foram sete: cor da flor, posição da flor no caule, cor da semente, textura da semente, forma da vagem, cor da vagem e altura da planta.[16]
Leis da Hereditariedade
As leis da hereditariedade proposta por Mendel são o resultado dos fundamentos que explicam a forma como ocorre a transmissão das características de geração para geração. Sendo elas, respectivamente, lei da segregação dos fatores (monoibridismo), lei da segregação independente (diibridismo) e lei da dominância.
A primeira lei, do monoibridismo, também recebe o nome Lei da Segregação dos Fatores, refere-se ao fato de que cada característica é composta por um par de fatores, no qual um fator advém da mãe e o outro do pai e ao se formar os gametas eles se separam com a mesma probabilidade, um exemplo dessa lei é o albinismo[17].
Mendel percebeu que plantas de sementes amarelas sempre produziam 100% dos seus descendentes com sementes amarelas. Após esse cruzamento, realizou a autofecundação de uma dessas sementes híbridas e o resultado encontrado da segunda geração foi 75% de sementes amarelas e 25% de sementes verdes. Com isso, Mendel concluiu que o fator para amarelo seria o dominante e o fator para verde recessivo.
Já a segunda lei, de diibridismo, estabelece uma formação dos gametas diferente do da primeira lei, nesse caso, os fatores se separam de forma independente na formação dos gametas, no entanto, os gametas produzidos seguem a mesma proporção [13].
Mendel realizou o cruzamento de plantas com diferentes características, cruzou plantas com sementes amarelas e lisas com plantas de sementes verdes e rugosas. Mendel já esperava que a geração F1 seria composta de por 100% das sementes amarelas e lisas, pois estas características apresentam caráter dominantes. [16].
Os genótipos e fenótipos cruzados eram os seguintes:
V_: Dominante (cor Amarela)
R_: Dominante (forma Lisa)
vv: Recessivo (cor Verde)
rr: Recessivo (forma Rugosa)
Na geração F2, Mendel observou diferentes fenótipos, nas seguintes proporções: 9 amarelas e lisas; 3 amarelas e rugosas; 3 verdes e lisas; 1 verde e rugosa.
A terceira lei acaba sendo uma junção das duas leis anteriores, ela assegura-se que o fator dominante se sobressai ao fator recessivo para aqueles que possuem descendentes com características diferentes, isto é, para os híbridos [18].
Genética Quantitativa
É o ramo da genética que estuda o caráter quantitativo dos seres, ou seja, se aprofunda no estudo das qualidades adquiridas pelos genes, enfatizando a herança e os fatores determinantes da variação genética. Esses estudos são baseados nas características quantitativas, na qual são controladas por vários genes, cada um com efeitos distintos e são muito influenciadas pelo ambiente, apresentando variações dentro das populações. Existem caracteres mais sensíveis que outros as diferenças ambientais. Um grande exemplo de característica quantitativa pode ser expressa pelo peso à uma determinada idade e até pela altura de uma determinada espécie de espiga de milho. A produção de grãos é bastante afetada pelo ambiente. Além disso, as taxas de ganho genético de algumas espécies que foram e ainda estão sendo obtidas em programas de melhoramento genético, demonstram claramente o poder do uso da genética quantitativa na seleção. Com isso, é fundamental o estudo integrado entre genética quantitativa e a genética molecular no estudo da variabilidade das características das diferentes espécies. [4]
Genética Molecular
A genética molecular é um ramo dentro da genética que procura entender como funcionam os genes a nível molecular, antes da genética molecular a investigação dos genes era feita a partir do fenótipo dos organismos estudados, mas, com a genética molecular pode-se entender como funcionam os genes e suas estruturas, fazendo assim o pensamento inverso a genética mendeliana, estudando os genes para poder prever as características que o organismo estudado vai receber, ou pelo menos suas possibilidades. [19].
É uma subdivisão do ramo da genética. Os genes são unidades de hereditariedade, instruções para características herdadas, como cor do cabelo, tipo sanguíneo e predisposição para algumas doenças. Este campo dentro das ciências biológicas preocupa-se com as propriedades físicas e químicas desses genes. Os organismos dependem das células para crescer e funcionar. Cada célula é essencialmente como uma máquina, seguindo as instruções de seus genes para funcionar com eficiência. A molécula que compõe os genes é o ácido desoxirribonucléico (DNA), que é armazenada dentro das células dos organismos. É uma molécula longa e firmada em estruturas chamadas cromossomos. Esses cromossomos requerem forte ampliação para serem visualizados. Quando vista desenrolada, a molécula de DNA se assemelha a uma escada retorcida, com duas camadas entrelaçadas chamadas hélice dupla. A estrutura da dupla hélice do DNA carrega grande parte da genética molecular. A genética molecular também se preocupa com a estrutura e a função do ácido ribonucleico (RNA), uma molécula essencial para o funcionamento das células. Em sua forma, o RNA é bastante semelhante ao DNA, mas eles têm funções diferentes. As informações no DNA da célula são copiadas para uma molécula de RNA, após a qual é transformada em uma proteína que será essencial para uma determinada tarefa.
Esta teoria diz respeito não apenas à composição, expressão e regulação dos genes, mas também ao papel geral dos genes dentro do organismo. De acordo com a teoria fundamental, genes e DNA direcionam todos os processos da vida, fornecendo as informações que especificam o desenvolvimento e o funcionamento dos organismos.
[15].
Genética de Populações e Evolutiva
A genética de populações busca estudar as frequências gênicas, genotípicas e fenotípicas nas populações e a distribuição e mudança na frequência de alelos sob a influência de quatro fatores evolutivos: seleção natural, deriva genética, mutação e o fluxo gênico, no qual são capazes de alterar as características ao longo das gerações das espécies. A frequência gênica expressa a proporção na população dos diferentes alelos de um gene e, a genotípica determina a porcentagem na população dos diferentes genótipos para o gene considerado. Este estudo indica a distribuição genotípica e a fenotípica da progênie resultante de todos os cruzamentos possíveis na população, tendo como base os fenômenos determinantes e como eles afetam a estrutura genética de uma população ideal. [20].
A evolução consiste na mudança gênica de uma população. Com isso, se uma população mantém constante sua frequência gênica ao passar das gerações é sinal de que ela não está em evolução, ou seja, não está sofrendo a ação de fatores evolutivos. Caso uma população tenha uma alteração da frequência gênica ao passar das gerações, é sinal que fatores evolutivos estão atuando sobre ela, assim, pode-se concluir que trata-se de uma população em processo evolutivo.
Por exemplo:
Geração 1: 30% de genes A e 70 % de genes a
Geração 2: 30% de genes A e 70% de genes a
Geração 3: 30% de genes A e 70% de genes a
Analisando as diferentes gerações, conclui-se que não há variação na frequência dos genes analisados (A e a), isso mostra que não há ocorrência de fatores evolutivos e, consequentemente, não ocorreria um processo de evolução desta população. Ela permanece em equilíbrio ao longo das gerações. Dessa forma, para determinar se uma população está em evolução ou não, é necessário levar em conta a frequência gênica ao longo das gerações e, por fim, concluir se houve variação.
Genética do Desenvolvimento
A genética do desenvolvimento se baseia no estudo dos mecanismos genéticos que promovem o desenvolvimento de um organismo. Ele envolve as mudanças no nível de funcionamento do indivíduo ao longo do tempo, uma vez que o desenvolvimento é um processo de mudança que dura a vida toda, seja de um animal, ser humano ou planta. O material genético dirige o crescimento e a diferenciação do organismo a partir do zigoto unicelular até o adulto. Para controlar esse processo, o material genético se expressa em cada gene que, deve agir no momento e no local preciso, de forma a garantir que o fígado, por exemplo, seja formado por células hepáticas, o sistema nervoso por células nervosas e assim por diante. É comumente assumido que o crescimento, a maturação e o desempenho físico de um organismo, são afetados pela hereditariedade. A herança biológica representa as influências da geração dos pais sobre a geração dos filhos, que são mediadas por genes. Uma influência ou efeito genético está associado a um gene ou um grupo de genes codificados no DNA dos cromossomos no núcleo das células. Existem um conjunto de genes, por exemplo, associados a estatura e ao peso, outros associados as dimensões ósseas e, com isso, os indivíduos diferem consideravelmente nos seus índices de maturação[21].
Conceitos de DNA, RNA e Proteínas
Estrutura do DNA e RNA
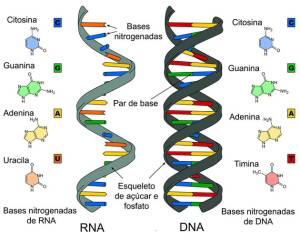
O ácido desoxirribonucleico (DNA) é uma molécula, formada por uma junção de átomos arranjados de forma espiral, composta por nucleotídeos que são as unidades básicas dos ácidos nucleicos. Sua estrutura é formada, basicamente, por duas fitas, constituídas de açúcar e fosfato, em formato de dupla hélice, que são conectadas, uma com a outra, por ligações de hidrogênio formando as bases nitrogenadas. As bases nitrogenadas, presentes no DNA, são construídas por compostos em pares, são eles: Citosina (C) e Guanina (G); Timina (T) e Adenina (A). O ácido ribonucleico (RNA), assim como o DNA, também é uma molécula formada por uma junção de átomos compostas por nucleotídeos, porém sua estrutura é formada por apenas uma fita constituída, também, de açúcar e fosfato. Além disso, o RNA possui, assim como o DNA, pares de bases nitrogenadas com a diferença de possuir Uracila (U) no lugar de Timina (T). Portanto, as bases nitrogenadas presentes no RNA são: Citosina (C) e Guanina (G); Uracila (U) e Adenina (A) [22][23][2][24].
Função do DNA
Os primeiros estudos de decifração da estrutura do DNA, segundo Embrapa, foi realizado por James D. Watson e Francis H. Crick quando, em 1953, publicaram na revista Nature o artigo A Structure for Deoxyribose Nucleic Acid, no qual os autores apresentaram, primariamente, o estudo sobre a estrutura do DNA. O estudo foi extremamente importante para a época e com base nele muitos outros estudos acerca do tema surgiram. Além disso, o estudo proporcionou, em 1962, um prêmio nobel de medicina para os autores[25].
De maneira geral, pode-se dizer que tanto o ácido desoxirribonucleico (DNA) e o quanto ácido ribonucleico (RNA) armazenam informações, porém trabalham de formas diferentes.
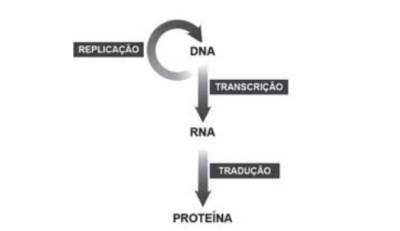
A principal função do DNA é armazenar e transmitir informações genéticas. A transmissão de informação genética se dá através de 3 grandes passos, são eles: Replicação, transcrição e tradução e o DNA está presente em dois deles, replicação e transcrição. Na replicação, que ocorre no núcleo das células, as fitas de DNA são copiadas de modo que as informações presentes em uma, continuam armazenadas nas fitas seguintes, sem que haja perda de informação. Após isso, ocorre o processo de transcrição que é quando as informações presentes nas moléculas de DNA, são copiadas para as moléculas de RNA, gerando um RNA mensageiro que será usado para a última etapa, tradução, que é o processo no qual os dados armazenados no RNA, oriundos no DNA, são traduzidos para dar origem a síntese proteica, que ocorre no ribossomos, e que é basicamente a produção de proteínas.[18][26][16].
Replicação do DNA
A replicação do DNA ocorre no processo de divisão celular, mais precisamente na interfase, duplicando uma molécula de DNA de dupla cadeia, como cada uma dessas contém a mesma informação genética, qualquer uma poderá servir como molde, por conta disso a replicação do material genético da célula é chamada de semi-conservativa. Os mecanismos de replicação dos procariontes e eucariontes são idênticos, sendo um processo essencial para o desenvolvimento e manutenção dos organismos assim como a formação de gametas para a propagação da hereditariedade. Porém, este processo é suscetível a erros, pois como a reprodução celular é realizada diversas vezes todos os dias, as chances de ocorrerem falhas no momento da replicação das fitas do DNA não são desprezíveis. Como as moléculas de DNA carregam em sí todas as informações genéticas de um indivíduos, com as mutações podem ocorrer sinteses de alelos errados ou até mesmo a não produção de alélos essenciais ou não para o organismo. Estas falhas, quando não “fatais” as moléculas e/ou células são passadas de geração em geração das moléculas de DNA.
Os motivos para mutações ocorrerem são diversos, em alguns casos expontâneas, ou seja, sem nenhum fator externo aos agentes celulares e de difícil atribuição, e em outros casos serem decorrentes da ações de agentes mutagenicos externos a estrutura da célula e do organismo. Suas naturezas podem ser químicas (bases análogas a bases nitrogenadas natívas ao DNA que se incorporam durante a síntese) ou físicas (radiações em geral). [27]
Iniciador- Molde
Uma nova síntese precisa dos 4 desoxinucleosideos trifosfatados (A,G,T,C). O segundo substrato essencial para a sínteses é um arranjo de DNA de fita simples e DNA dupla-fita chamado junção iniciador:molde. O molde fornece a fita simples que dirige a adição de cada desoxinucleotideo complementar. O iniciador é um segmento curto de DNA complementar ao molde. “O iniciador deve possuir um grupo 3'-OH adjacente exposto para a região de fita simples pois é neste grupo que sera estendido a medida que os nucleotídeos forem adicionados.” [28]
As bases químicas da síntese de DNA permitem que a extensão da nova cadeia ocorra apenas pela extremidade 3' do iniciador. A fita molde orienta qual dos quatro nucleosídeos trifosfatados será adicionado.
Mecanismos da DNA – Polimerase
A síntese do DNA é catalisada por uma classe de enzimas chamada DNA - Polimerase, elas adicionam nucleotídeos um por um, somente na terminação 3' da fita crescente de DNA, incorporando somente aqueles que são complementares a fita molde. Essa enzima utiliza um único sitio ativo para catalisar a adição de qualquer um dos 4 desoxinucleosideos trifosfatados. “Em vez de detectar o nucleotídeo exato que entra no sitio ativo, a DNA- polimerase monitora a capacidade de o nucleotídeo a ser incorporado formar um par de bases A:T ou G:C . Apenas quando um par de bases correto é formado, o grupo 3'-OH do iniciador a o grupo alfa fosfatado do nucleosídeo trifosfatado estão na posição correta”[29], se inicia o processo de replicação.
Exonucleases - Revisão de leitura no DNA
“São enzimas que atuam na clivagem de nucleotídeos a partir do final de uma cadeia polinucleótica, eliminando um nucleotídeo por vez. Isso ocorre por uma reação de hidrolise que que ligações fosfofdiester pela extremidade 3' ou 5'. Elas também removem nucleotídeos mal pareados, permitindo assim que pares de bases incorretas sejam posicionadas corretamente e e posteriormente sejam eliminadas”.[30] “A remoção desses nucleotídeos malparados é medida por uma nuclease originalmente identificada no mesmo polipeptídeo. Chamadas de exonucleases de revisão de leitura.”[31]
Embora a exonuclease somente atue em extremidades de uma fita de DNA, há uma outra enzima que consegue clivar no meio de uma fita, elas são chamadas de endonucleases.
Forquilha de Replicação
Quando ocorre a replicação do DNA, as duas fitas do duplex de DNA estão sendo replicadas ao mesmo tempo, para isso é necessário que essas fitas fiquem separadas a fim de evitar que elas se juntem novamente. A junção entre a fita molde e a recém formada é feita pela forquilha de replicação, se trata de uma estrutura formada no núcleo durante o processo de replicação do DNA, esta é criada pela helicase, que quebra as ligações de hidrogênio responsáveis por ligar as duas cadeias de DNA, a forquilha se desloca em direção a região do duplex ainda não replicado de DNA, deixando dois moldes de fita simples que replicam cada.
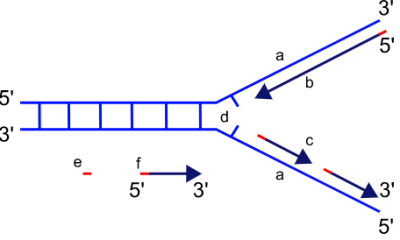
A estrutura resultante deste tem duas ramificações, cada uma formada por uma cadeia de DNA, são chamadas de fita líder e fita atrasada. O DNA tem uma natura antiparalela, ou seja, ela não consegue replicar simultaneamente os dois moldes expostos pela forquilha, então a medida que a forquilha se movimenta, somente uma dessas fitas pode ser replicada de forma continua, damos o nome de fita líder para essa fita.
A síntese da outra fita é feita enquanto a DNA polimerase se desloca na direção oposta a forquilha, recebendo o nome de fita atrasada.
“A DNA - polimerase na fita líder pode sintetizar o DNA assim que seu molde for exposto,porem, a síntese da fita tardia precisa esperar que o deslocamento da forquilha de replicação exponha uma extensão considerável do molde antes que este possa ser replicado”.[25]}}]
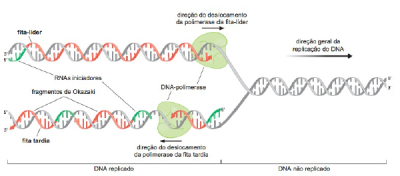
Os chamados Fragmentos de Okazaki são pequenas frações de DNA recém sintetizados que foram formados na fita tardia e após sua síntese, estes são ligados covalentemente produzindo uma nova fita de DNA. São portanto intermediários temporários na replicação do DNA.
Iniciação de uma nova fita de DNA
As DNA - polimerases precisam de um iniciador, como dito anteriormente e para isso a célula inicia novas cadeias de RNA. A capacidade de sintetizar esses iniciadores é responsável pela primase (que é uma RNA - polimerase). “Embora as DNA - polimerase incorporem somente desoxirribonucleotideos no DNA, elas podem iniciar a síntese utilizando um iniciador de RNA ou de DNA anela ao molde de DNA”.[34]
A primase funciona de forma diferente depende de qual fita esta atuando (fita-inicial ou fita-tardia). Enquanto que a fita-líder precisa apenas de um único iniciador de RNA, a fita tardia precisa da síntese de um novo iniciador a cada fragmento de Okazaki. Diferentemente das sínteses de RNA mensageiro, RNA ribossômico e RNA transportador as primases iniciam a síntese de RNA usando um trímero especifico. Ao se associar a proteína DNA - helicase, a atividade da primase é aumentada. Isso acontece pois a helicase desenrola o DNA na forquilha criando um molde de fita simples onde a primase consegue atuar.
Remoção dos iniciadores de RNA
Para fazer essa substituição, uma enzima denominada RNase H reconhece e remove a maior parte de de cada indicador de RNA, degradando especificamente o RNA que esta realizando o pareamento de bases com DNA. A RNase H remove todo o indicador de RNA exceto o ribonucleotídeo diretamente ligado a extremidade do DNA (a RNase H só consegue clivar ligações entre dois ribonucleotídeos). A remoção do RNA deixa uma lacuna no DNA dupla fita funcionando como um substrato ideal para a DNA - polimerase. Essa, preenche a lacuna por pareamento de cada nucleotídeo, deixando uma molécula de DNA completa exceto por uma quebra que é reparada por uma enzima chamada DNA - ligase.
Separação moléculas - filha DNA
Após o término da replicação, as moléculas - filhas permanecem ligadas ou concatenadas. Para separar essas moléculas, elas precisam ser desentrelaçadas e para isso enzimas de topoisomerases de tipo II começam a atuar pois as mesmas conseguem quebrar uma molécula de fita dupla e passar uma segunda molécula de fita dupla através dessa quebra.
Mutações
Podem ocorrer em células somáticas ou germinativas em organismos multicelulares. Quando ocorrem em células somáticas, estas mutações são as maiores responsáveis pela origem de câncer em indivíduos, e portanto, sendo uma das maiores causas das mortes no mundo moderno. Porém nem sempre estas mutações evoluem para tipos de câncer ou outros tipos de consequências genéticas graves aos indivíduos. Como ocorrem em células estruturais não “transmissíveis” a outros indivíduos, as mutações em células somáticas permanecem no próprio indivíduo, a não ser que haja reprodução assexuada.
Já as mutações em células germinativas são aquelas que podem influenciar na carga genética de um indivíduo originado destas células. Como estas mutações são aleatórias não há como prevê-las, ou seja, o mecanismo de seleção natural será encarregado de eliminar ou (em poucos casos) perpetuar esta mutação que pode ser benéfica aos indivíduos portadores. [35]
Função do RNA
O RNA é responsável, como vimos anteriormente, pela transmissão das informações presentes no DNA para que seja feita a produção de proteínas. Porém, diferentemente do DNA essa molécula pode ser subdividida em três classes: RNA mensageiro, RNA transportador e RNA ribossômico. O RNA mensageiro é a cópia do DNA, que ocorre na transcrição. O RNA transportador possui a função de transportar os aminoácidos, que serão usados na construção de proteínas, para os ribossomos e, por fim, os RNA ribossômico que são localizados, como o próprio nome diz, no ribossomo e que quando são sintetizados se aglomeram para formar os nucléolos que se combinam com as proteínas[16][17].
Além disso existem os catalisadores das reações da síntese do RNA, conhecidos como RNA polimerase (ARN-polimerase). São enzimas que atuam no processo de tradução e por fim auxiliam também no processo estrutural e regulador, podendo até ser sintetizado uma nova cadeia de RNA, onde apenas um molde do DNA é necessário, não necessitando de iniciador.
Íntrons e Éxons
Íntrons e éxons tratam-se de sequências de nucleotídeos dentro de um gene, onde os íntrons funcionam como sequência de intervenção e os éxons como sequências expressadas. Quando uma molécula de RNA é sintetizada é chamada de transcrito primário, que nem sempre são funcionais. Os transcritos primários para um mRNA também são chamados de pré-mRNA. Em células eucariontes, os transcritos eucariontes possuem uma diferença marcante em relação aos procariontes, pois são bem maiores do que os transcritos primários esperados para proteínas de procariontes. Na maioria dos genes estruturais eucarióticos, as sequências codificantes são espaçadas por regiões não expressas, os íntrons, que inicialmente são transcritas em RNA no núcleo, enquanto que as regiões codificadas são os éxons. Um pré-mRNA, geralmente, contém oito íntrons. Quando falamos da questão de tamanho cumulativo dos éxons e dos íntrons no gene, os éxons tendem a ser muito menores que os íntrons. O mRNA maduro surge apenas após a conclusão da remoção dos íntrons e da poliadenilação do RNA. O mRNA é transportado para o citosol, onde se localizam os ribossomos, para que ocorra a tradução em proteína[36] [37].
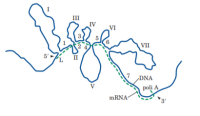
A descoberta de que nem todos os genes eram contínuos foi feita em 1977, por Phillip Sharp e Richard Roberts quando os dois cientistas prepararam amostras de mRNA que podiam hibridizar com o DNA e visualizaram o resultado, onde notaram que uma única molécula de mRNA hibridizou com, no mínimo, 4 segmentos separados do DNA, onde as sequências que não foram pareadas formaram alças. Na figura ao lado, é possível observar as posições dos éxons e as posições dos íntrons, estes que são os segmentos que formam as alças para fora e não possuem regiões complementares no mRNA [36] [37].
Tipos de Íntrons
Existem quatro tipo de íntrons, sendo que as duas primeiras classes, grupo I e grupo II, se diferem em relação aos detalhes dos seus mecanismos de splicing, porém, ambos possuem a característica de realizar auto splicing.
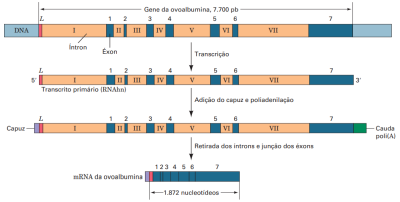
Durante o processo de splicing, os íntrons são removidas da transição e os éxons são ligados para formar uma sequência contínua de um polipeptídeo funcional [36].
Grupo I
O grupo I são encontrados em genes nucleares, mitocondriais e de cloroplastos quem codificam rRNAs, mRNAs e tRNAs. Durante o splicing deste grupo, é necessário de um nucleotídeo guanina ou de um cofator nucleotídeo. O grupo da guanina é utilizado como nucleófilo na primeira etapada do splicing. Na segunda etapa, a hidroxila do éxon é deslocada e age como nucleófilo em um reação com o íntron. O resultado deste processo é a remoção do íntron e a ligação dos éxons [37].
Grupo II
O grupo II geralmente é encontrado em mRNA mitocôndrias, cloroplastos em fungos, algas e plantas. No grupo II, a diferença está no nucleófilo da primeira etapa, neste caso a hidroxila do íntron pertence a outro grupo [36][37].
Íntrons do spliceossomo
Este grupo não sofre auto splicing e é encontrada em mRNA, é a maior classe dos íntrons. Possuem esse nome pois sua remoção é catalisada por um complexo protéico chamado de spliceossomo [36].
Quarta classe dos íntrons
A quarta classe é encontrada em alguns tRNA e se distingue dos demais grupos pelo fato da reação de splicing precisar de ATP. Durante o splicing, ocorre a separação em ambas as extremidades dos íntrons e os dois éxons são ligados por uma mecanismo de reação DNA-ligase [36].
Éxons
Os éxons são sequências codificantes dos genes, essas são intercaladas por regiões não codificantes, os chamados íntrons. Em sua maioria o tamanho cumulativo dos exons é muito menor que o de íntrons, contendo cerca de 150 nucleotídeos, enquanto os íntrons podem possuir várias centenas de nucleotídeos. Essa desproporção entre exons e introns aumenta a probabilidade de recombinação, com maior frequência nos íntrons que nos éxons em eucariotos superiores.
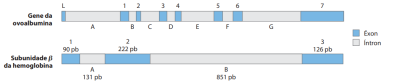
Para a expressão correta, um gene também inclui as sequências adjacentes necessárias, na sua quantidade correta, no local correto e no momento correto do ciclo celular, para que assim seja um gene com função específica deste.
As sequências adjacentes são as que indicam o início e o fim da transcrição do gene. A região de início está na região promotora e é chamada de extremidade 5’, enquanto a chamada extremidade 3’ é a região de término.
Óperons
Os óperons são unidades genéticas pertencentes a genomas procarióticos, onde os genes são arranjados lado a lado ao longo de uma única fita de DNA e transcritos juntos.[36]A maioria dos genes bacterianos são reunidos e regulados em óperons, uma vez que em seu mecanismo para coordenar e regular os genes que codificam produtos que participam em um conjunto de processos relacionados, os genes se mantém agrupados no cromossomo e são transcritos juntos. Em muitos casos, no mRNA das bactérias, há múltiplos genes em um único transcrito, sendo o local de regulação para a expressão de todos os genes no grupo o único promotor que inicia a transcrição do grupo de genes, onde os óperons serão esse grupo de genes e o promotor, mais as sequências adicionais que funcionam associadamente na regulação.
O metabolismo da lactose presente em bactérias E. coli desencadeou estudos voltados à expressão dos genes bacterianos, já que essa bactérias conseguem utilizar a lactose como sua única e exclusiva fonte de carbono. François Jacob e Jacques Monod (Nobel de Medicina em 1965), estudiosos franceses, por volta dos anos 1960, por meio de um artigo publicado no jornal Proceedings, trouxera à tona seus estudos sobre a regulação de dois genes adjacentes presentes no metabolismo da lactose por um outro elemento genético, que por sua vez se encontrava na extremidade do grupo de genes. Tratava-se da 𝛽-galactosidase, enzima responsável pela quebra da lactose em galactose e glicose e, também, da lactose-permease, responsável pelo transporte da lactose para o interior da célula. Foi esse artigo que trouxe pela primeira vez os termos “óperon” e “operador”, que elevou a regulação gênica à termos moleculares.[36]
Óperon lac sujeito à regulação negativa
O óperon lactose (ou óperon lac) inclui os genes para β-galactosidase (Z), galactosídeo-permease (Y) e tiogalactosídeo-transacetilase (A) e cada um dos três genes é precedido por um sítio de ligação de ribossomos que direcionam a tradução de determinado gene [36].
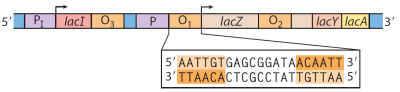
Através de estudos foi possível observar que existe uma proteína responsável por reprimir (inibir) a transcrição do óperon lac, é chamada de repressor Lac, um tetrâmero de monômeros idênticos e se liga mais fortemente ao operador O1, que por sua vez é adjacente do sítio de início de transcrição. Existem mais dois sítios de ligações secundários para o repressor Lac, são eles o O2, centralizado próximo da posição +410 e o O3, próximo da posição -90. Dessa forma, para reprimir o óperon, o repressor pode-se ligar em qualquer um dos sítios mencionados, bloqueando a iniciação da transcrição [36].
Ressalta-se que a ligação do repressor Lac reduz a velocidade de transcrição em 10^3 vezes, além disso se os sítios O2 e O3 forem eliminados, somente a ligação com o sítio O1 seria responsável por diminuir a velocidade em 10^2 vezes. Por fim, é importante mencionar que o indutor no sistema óperon lac não é a própria lactose e sim a alolactose, um isômero de lactose, que é convertido por meio das poucas moléculas existentes de β-galactosidase [36].
Óperon lac sujeito à regulação positiva
Às regulação dos Óperons não são simples, já que o ambiente de uma bactéria é complexo o suficiente para que seus genes sejam controlados apenas por um sinal. Como a lactose, a disponibilidade de glicose também é um fator que afeta a expressão dos genes lac. Quando a metabolização é feita diretamente pela glicólise, que é a fonte de energia preferida em E. coli. Lembrando que outros açúcares também podem ser usados como único nutriente principal, mas para isso, é necessário prepará-las, logo o uso de sínteses adicionais. Por isso, o uso de outros açúcares ,como a lactose ou até mesmo arabionose, para expressar os genes para proteínas é um desperdício, visto que a glicose é abundante.
Quando a glicose e a lactose estão presentes no operon Lac, um mecanismo com função regulatória designado como “repressão catabolitos” que restringe a expressão dos genes fundamentais para catabolismo da lactose, mesmo quando existe açúcares presentes na glicose. O efeito da presença da glicose é medido através do cAMP, sendo coativador e uma proteína com funções ativadoras, denominada como proteína receptora de cAMP ou CRP,as vezes chamada de CAP (proteína ativadora de catabólito) .O CRP é uma homadímero com sitios de ligações para o DNA e o cAMP. Na ausência de glicose, a CRP-cAMP se liga a um sítio próximo da Lac e estimula a transcrição do RNA em um valor de 50 vezes, logo CRP-cAMP é um elemento regulatório positivo, que se baseia nas respostas conforme o nível de glicose e o repressor Lac é um elemento regulatório negativo que responde à lactose, ambos atuam em conjunto. A glicose no CRP tem seu efeito mediado pela interação com a cAMP. A CRP se liga ao DNA mais facilmente quando há altas concentrações de cAMP. Conforme a cAMP declina, a CRP ligado ao DNA diminui, o que reduz a expressão do óperon lac [36].
Uma rede de óperons com um regulador comum é denomiado regulon, e são estes que envolvem a CRP e a cAMP.
Semelhanças e Diferenças do DNA e RNA
Apesar do ácido desoxirribonucleico ser parecido com o ácido ribonucleico em muitos aspectos é possível encontrar algumas características peculiares ao DNA diferentes do RNA. Por exemplo, o DNA, como vimos anteriormente, é um material genético que apresenta como estrutura uma fita dupla hélice e é composto pela pentose desoxirribose, grupo fosfato e quatro bases nitrogenadas: adenina (A), citosina (C), guanina (G) e timina (T) (4(2009)) enquanto a molécula de RNA apresenta como estrutura uma única fita simples que também é composta por uma pentose, porém diferente da presente no DNA pois, no RNA encontramos a ribose. Além disso, o RNA é composto por um fosfato e quatro bases nitrogenadas, sendo elas: a adenina (A), guanina (G), citosina (C) e a uracila (U). Nota-se que no lugar da timina (T) do DNA entra a uracila, no RNA[38] [39] [17][40].
Proteína
A proteína é considerada quimicamente, um dos compostos mais complexos e com maior aprimoramento que conhecemos. Compõe mais de cinquenta por cento da massa seca de uma célula e sua síntese tem uma importância fundamental para a manutenção e o crescimento das células. Esta síntese é realizada no ribossomo e envolve vários tipos de moléculas de RNA que atuam nas diversas etapas do processo. [7] [41].
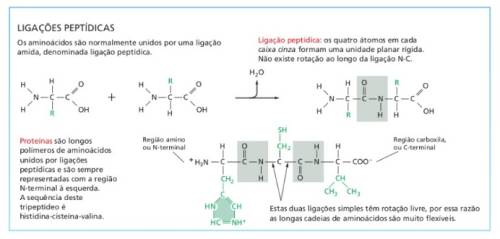
A proteína é formada por aminoácidos conectados por ligação peptídica, e para entender melhor sobre proteína/polipeptídeos é importante discorrermos sobre aminoácidos. São vinte tipos de aminoácidos que foram divididos aqui em cadeias: Aminoácidos de cadeia Lateral alifática: glicina, leucina, isoleucina, alanina, Valina, Metionina e Prolina; Aminoácidos de cadeia Lateral Aromática: Fenilalanina, Tirosina e Triptofano; Aminoácidos de cadeia Lateral hidrofóbica : Serina, Glutamina, Treonina, Cisteína e Asparagina; Aminoácidos de cadeia Lateral Carregada: Lisina, Arginina, Histidina, Aspartato, Glutamato.[42].
As quantidades diferentes de aminoácidos geram proteínas diferentes entre si, é importante ressaltar que para um conjunto de aminoácidos serem considerados proteínas, esse conjunto deve ter no mínimo uma combinação de cem aminoácidos e os chamados Polipeptídios tem no mínimo 50 aminoácidos.[32]
Além de que proteínas são, quimicamente, uma cadeia principal polipeptídica ligada a duas cadeias laterais, logo as diferentes sequência de cadeias laterais geram diferentes proteínas também[7].
Níveis Estruturais de Proteínas
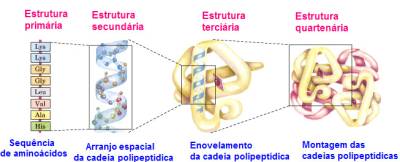
Existem basicamente quatro níveis estruturais. O primeiro é uma sequência de aminoácidos que são unidos por ligações peptídicas (onde a substituição de um aminoácido por outro pode causar doenças), e todas as outras estruturas dependem dessa primária. Já as estruturas secundárias, sendo as principais hélices-e folha-, são basicamente primárias que sofreram ligações de hidrogênio (H) entre os aminoácidos -NH e C=O que fizeram com que ficassem em formato helicoidal. Essas estruturas também são caracterizadas por padrões regulares e repetitivos, que acontecem devido à atração entre alguns átomos de aminoácidos próximos. Já a estrutura terciária, que corresponde ao dobramento da cadeia polipeptídica sobre ela mesma, segue todos os pontos do enovelamento tridimensional (3D) de um polipeptídio. E por fim a quaternária, é quando duas ou mais estruturas terciárias (cadeias polipeptídicas idênticas ou não) se juntam para formar a estrutura total da proteína[32][7].
Síntese Proteica
O processo da Síntese proteica consiste em produzir proteínas, este ocorre nos ribossomos das células procarióticas e eucarióticas. Na síntese proteica as células produzem proteínas, determinado pelo DNA. Pode-se afirmar também que é um processo biológico no qual as informações que estão presente no DNA da célula é transformado em proteínas, que são uma das macromoléculas fundamentais para a subsistência do organismo, essencial para que ocorra a manutenção e o crescimento celular, chamada “tradução” de informação genética, podendo ser nomeada também como tradução gênica.
As proteínas são consideradas essenciais para o nosso organismo e podem ser formadas por um ou mais polipeptídios, a união de aminoácidos acontece mediante a sequência de códons do RNA mensageiro (RNAm). Sendo que a sequência desses códons é definida pela sequência de bases nitrogenadas do DNA[18].
As fases da síntese proteica entendem o DNA “transcrito” pelo RNA mensageiro (RNAm). Sendo assim, a informação transcrita será traduzida pelos ribossomos e pelo RNA transportador (RNAt) que transportará aminoácidos, em que a sequência irá determinar a formação da proteína. [44]
São encontradas no espaço intracelular da síntese de proteínas:
• Ribossomos livres no citosol sintetizam proteínas que serão utilizadas posteriormente a esse processo no citosol. Já as proteínas que possuem pontes dissulfeto não podem ser sintetizadas por essa subpopulação, pois o citosol é um ambiente redutor
• “Ribossomos associados ao retículo endoplasmático sintetizam proteínas que serão exportadas para o meio extracelular, direcionadas a outras organelas ou inseridas em membrana. Nesse caso, as proteínas são “internalizadas” no retículo concomitantemente ao processo de tradução”[45].
A síntese proteica é constituída por em três fases, sendo elas:
Iniciação da tradução: Nesta primeira fase há a união das duas subunidades do ribossomo com o RNAm (RNA mensageiro) e RNAt (RNA transportador), levando o primeiro aminoácido da cadeia polipeptídica.
Alongamento da cadeia polipeptídica: Nesta segunda fase , os outros aminoácidos que fazem parte da cadeia polipeptídica são acrescentados. O anticódon do RNAt se emparelha com o RNAm no sítio A. O RNAr catalisa a formação da ligação peptídica entre o novo aminoácido e a cadeia em formação. Assim o polipeptídio é desagregado do RNAt que está no sítio P e conectado ao aminoácido do RNAt do sítio A. O RNAt que está no sítio P é deslocado ao sítio E e retirado, em seguida, do ribossomo, enquanto o RNAt do sítio A é deslocado ao sítio P. O RNAm também é deslocado no ribossomo e leva para o sítio A o próximo códon a ser traduzido, assim é dada sequência ao processo até a identificação do códon de término.
Término da tradução: Nesta terceira e última fase, posterior a fase a identificação do códon de término, há uma proteína, a de fator de término, que se conecta ao códon induzindo a ligação da molécula de água na porção final da cadeia, fazendo com que ocorra a ruptura da ligação entre o peptídeo e o RNAt presente no sítio P. O peptídeo formado é liberado por meio do túnel de término presente na subunidade maior do ribossomo.
Ao fim do processo descrito, as cadeias polipeptídicas geradas podem passar por diferentes processos de transformação, assim tornando as proteínas funcionais[46][47].
Transcrição e Transcrição Reversa
Para que as informações genéticas possam ser passadas para uma molécula de RNAm ( tanto para um eucariota quanto para um procariota), as mesmas precisam ser transcrita para posteriormente serem traduzidas para um polipeptídeo funcional. Umas das diferenças nesse processos em relação as Procariontes é que todo os RNA são produzidos por um único RNA polimerase enquanto nas Eucariotas encontramos 3 tipos de RNA polimerases l, ll e lll.
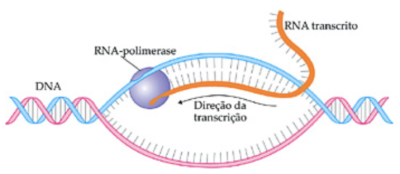
Portanto transcrição, em resumo, é a função de produzir qualquer tipo de RNA (m, t, r), que por sua vez, também pode ser chamado de transcrito. Esse processo se baseia na replicação de uma das fitas do DNA que é usado como molde, contudo, nem todo DNA é transcrito somente os genes nos quais codificam o novo RNA (não apresenta primase, helicase, e ligase). Todo o procedimento fica em função do RNA polimerase, mas quando todas as enzimas se agrupam sobre o DNA, numa condição chamada de RNA polimerase holoenzima. Uma questão importante é que a RNA polimerase copia uma das fitas do DNA, a fita molde, e assim o novo RNA é igual a fita não molde (lembrando que o RNA possui uracilas ao invés de timinas fazendo assim o chamado codificante). Mesmo ocorrendo a separação de cadeias no DNA, nao e o bastante para o inicio da sintense de RNA. Devido a isso, a polimerase acessa a sequencia por meio das alças e a unidade σ acaba sendo extremamente importante nesse reconhecimento tendo acesso a 12 pares de bases. Apos isso as cadeias devem ser separadas para permitir o pareamento e isso resulta nas ligações ionicas entre aminoácidos e o fosfato acídicos[48].
Os estágios presentes na transcrição são iniciação, alongamento e término sendo bem distintas em eucariotas e procariotas. Nos eucariotas a transcrição se inicia bem no início do filamento devido a fatores gerais de transcrição (pequenas partículas proteicas que auxiliam no reconhecimento e afinidade da RNA polimerase ao promotor), feita pela RNA polimerase ll. No alongamento tanto em bactérias quanto em eucariotas, há a bolha de transcrição que expõem o filamento molde (splicing ou recomposição) , porém para eucariotas é o momento em que o RNAm passa pela bola para receber a transcrição e posteriormente ser levado ao citoplasma para serem traduzidos. [49]
Os vírus só conseguem realizar o processo de reprodução dentro de uma célula viva (hospedeira). A replicação se inicia com o contato da membrana citoplasmática do hospedeiro com o envelope viral, ocorrendo a liberação do nucleocapsídio no citoplasma. No trânsito desse nucleocapsídio para o núcleo ocorre a transcrição reversa. Nesse processo o RNA viral serve de molde para o DNA simples, e após isso é degradado. Após isso, o DNA simples que foi sintetizado serve de molde para o surgimento de um outro DNA, a partir da formação de um LTR (Long Terminal Repeat) que flanqueiam o esse DNA, agora dupla fita.[50]
A transcrição reversa resulta em uma enzima, denominada de DNA-polimerase ou RNA-dependente, que é responsável por realizar o processo de transcrição, polimerizando moléculas de DNA a partir de moléculas de RNA, o que seria o contrário do que ocorre normalmente.
Como por exemplo o vírus do HIV, e outros semelhantes, são chamados de retrovírus. Já que por agirem de forma reversa, eles injetam seu material genético na célula que hospedam através da enzima e utilizam-se dos nucleotídeos presente na mesma para transcrever uma fita
Marcadores Moleculares
Marcadores genéticos são caracteres com mecanismo de herança simples que podem ser empregados para avaliar diferenças genéticas entre dois ou mais indivíduos. Estes marcadores podem, por sua vez, serem divididos em dois grupos básicos: marcadores morfológicos e marcadores moleculares onde, o primeiro é utilizado para identificação do genótipo e o segundo,por sua vez, é utilizado para detectar mutações que ocorrem no DNA por exemplo, e, permitir que o mesmo seja explorado [51].
A descoberta da mutação do polimorfismos de DNA mostra um exemplo da importância dos marcadores moleculares onde, marcadores de DNA podem ser divididos em três categorias principais: os baseados em hibridização, os baseados em PCR R (Reação em cadeia da Polimerase – Polymerase Chain Reaction) e por fim, marcadores baseados em sequenciamento [52]. Outro fator importante dos marcadores genéticos que vale a pena se pontuado é que os marcadores genéticos permitem que a seleção e novos cruzamentos de uma nova geração ocorra numa mesma geração, um relevante aspecto a ser considerado no melhoramento genético das plantas uma vez que, aumenta-se consideravelmente a eficiência de um programa de melhoramento [53].
Baseado em hibridização, o RFLP (Restriction Fragment Length Polymorphisms) é o primeiro marcador desenvolvido e servirão de base para os primeiros mapas genéticos. Com o DNA uma vez obtido, ocorre a clivagem com enzimas conhecidas, resultando em um grande número de fragmentos distintos. Estes fragmentos são separados, aparecendo as bandas de diferentes tamanhos, devido as substituições de base única. Trata-se de marcadores com expressão codominante e com resultados estáveis, porém possuem limitações quando se trata em automatizar suas etapas. Mesmo assim, são utilizados em estudos de variabilidade genética e identificação de espécies.
Os baseados em reação a cadeia da polimerase são 4: RAPD (Randomly amplified polymorphic DNA), AFLP (Amplified Fragment Lenght Polymorphism), ISSR (Inter Sequence Simple Repeats) e Microssatélites SSR (Simple Sequence Repeat). A técnica foi desenvolvida na década de 80 e apresentou uma nova opção no uso dos marcadores moleculares, sendo utilizada para ampliar pequenas sequencias especificas de nucleotídeos. É baseado na síntese enzimática de uma parte do DNA na presença da enzima DNA Polimerase, resultando em milhões de cópias. Ao comparar com RFLP citado no paragrafo anterior, nos RAPDs por exemplo, nota-se que necessitam cerca de 100 vezes menos DNA no processo, além de poderem ser observados diretamente no gel. Outra vantagem pode ser encontrada ao comparar RFLP com SSR, neste caso os baseados em PCR elimina a necessidade de cruzamentos distantes, deixando qualquer cruzamento informativo para o mapeamento molecular.
Por fim, os baseados em sequenciamento são SNP (Single Nucleotide Polymorphisms) e DArT (Diversity array technology). O primeiro pode ser considerado como os marcadores mais atraentes para a genotipagem, pois permite genotipar milhares de loci simultaneamente, além de serem fundamentais nas análises de genes, sua desvantagem é serem limitados para estudos de diversidade genética, além do alto custo. Já nos DArT, para detectar loci não há necessidade de uma sequência de informações. Além disso, uma vez descoberta um marcador, não é necessário ensaios de genotipagem específicos. Possuem alto rendimento e também são amplamente utilizados para genotipagem, carregando um alto custo. [54].